Cette plateforme propose des méthodologies, équipements et expertises pour la sélection, la caractérisation et l'ingénierie d'aptamères ADN, ARN ou acides nucléiques modifiés chimiquement pour résister aux nucléases.
La plateforme est ouverte aux industriels et aux laboratoires académiques pour des prestations ou des collaborations. En s'assurant de la protection des travaux (brevets, accord de confidentialité, MTA...), nous travaillons en étroite relation avec nos partenaires afin d'être au plus proche de leurs besoins. Il est possible d'identifier des aptamères contre tout type de cible (petites molécules, peptides, protéines, cellules...) et de les caractériser en terme d'affinité, de spécificité et d'activité biologique. Il est aussi possible de conjuguer l'aptamère avec un fluorophore ou molécule radioactive pour l'utiliser en imagerie.
Récemment, la plateforme a développé une méthode (PATTERNITY.seq©) qui permet d'utiliser le séquençage à très haut débit pour étudier simultanément plusieurs millions de séquences au cours d'un processus d'évolution. Cette analyse de "Big DATA" permet d'analyser les mécanismes d'évolution moléculaire avec une résolution inégalée. Elle permet de mieux comprendre l'effet des pressions de sélection et d'identifier de nouveaux aptamères avec des propriétés nettement améliorées.
Activités
- Synthèse, purification et marquage d'aptamères
- Sélection d'aptamères semi-automatisée
- Séquençage NGS et prédiction d'aptamères basée sur la méthode (PATTERNITY.seq©)
- Ingénierie d'aptamère (réduction de taille, modifications chimiques, marquage fluorescent et radioactif)
- Caractérisation d'affinité (Kd, Kon, Koff, Cmax) et d'internalisation cellulaire
- Evaluation in vivo de l'effet biologique de ces aptamères avec possibilité de prise en charge par la plateforme d'imagerie (imagerie optique et TEP) (au cas par cas)
Automate pour la sélection et la caractérisation d'aptamères
| Amélioration de l’affinité d’un aptamère pour des cellules MCF-7 suite à une ingénierie moléculaire. En haut affinité de l’aptamère de départ, en bas affinité de l’aptamère amélioré.
|
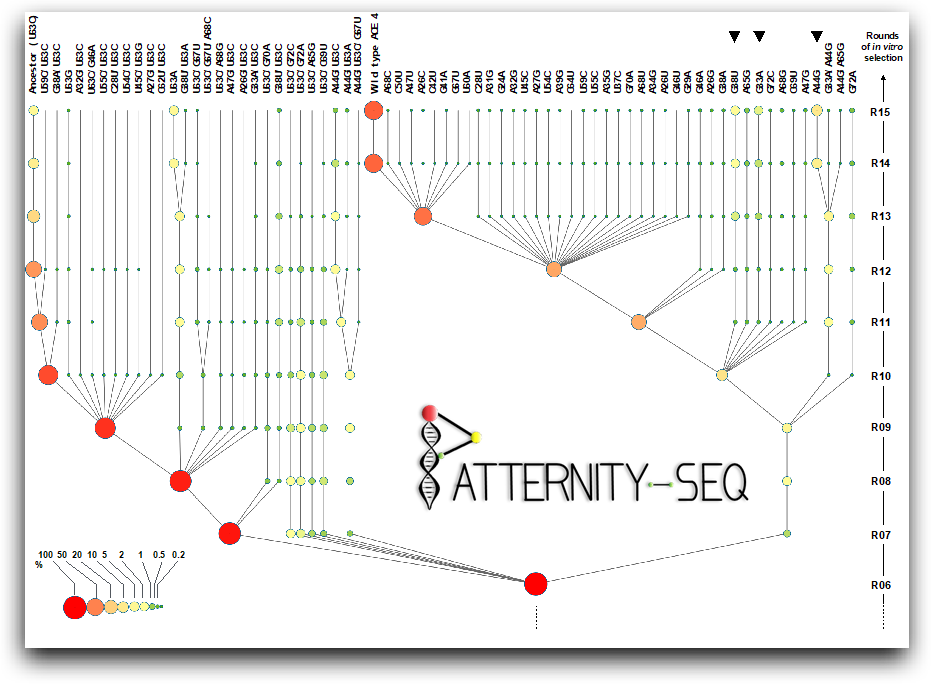
Suivi par séquençage à haut-débit de l'évolution des variants d'un aptamère au cours d'un SELEX
|
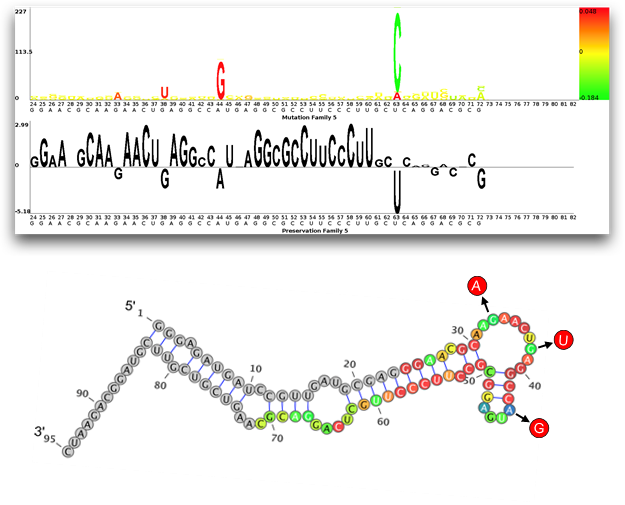
Analyse par séquençage haut-débit des mutations pouvant améliorer l’affinité d’un aptamère
|
Equipements
- Plateforme semi-automatisée pour la sélection d'aptamères.
- Equipement de biologie moléculaire pour la production et la purification d'aptamères
- Salles de culture de niveau de biosécurité 2
- Salle dédiée à la manipulation de radioéléments et à la mesure d'affinité (Automate, LigandTracer).
- Séquenceur NGS Illumina
- Station de travail pour analyse bioinformatique
- Système d'imagerie par fluorescence proche infra-rouge équipé de deux lasers (680 et 740nm) permettant une imagerie quantitative en 3D par tomographie optique de diffusion (fDOT) à une résolution du mm3
- Système d'endo-microscopie confocale fibrée (pCLE) équipé d'un laser à 488nm et permettant l'imagerie in vivo de sondes fluorescentes à une résolution de quelques microns