A la suite d’un inventaire approfondi des espèces décrites dans la littérature, il nous est apparu que l’espèce
Acinetobacter baylyi, isolée du sol, présente des avantages réels pour la considérer comme un modèle d’étude de métabolisme bactérien.
A. baylyi est le microorganisme doté de la capacité de transformation par de l’ADN exogène la plus élevée (compétence (1) naturelle). Cette propriété associée à sa capacité à faire de la recombinaison homologue (2), rend les modifications génétiques très aisées. C’est un processus d’échange ciblé (mutagénèse dirigée) qui permet de modifier spécifiquement une information génétique en un locus donné.
A. baylyi fait partie des gamma protéobactéries comme
E. coli avec qui elle partage 46% des gènes. De plus elle possède quelques avantages décisifs sur
E. coli : compétence naturelle, croissance plus rapide sur milieu minimum et pas d’ambigüité métabolique (aérobie stricte).
Mise en place de ressources pour l’étude systématique du génome d’A. baylyi
Séquençage et annotation du génome d’A. baylyi
Afin de disposer d’un modèle génétique plus manipulable qu’E. coli, nous donc avons entrepris un travail d’analyse systématique du génome d’A. baylyi en vue d’approfondir et de compléter la connaissance des fonctions métaboliques. Dans ce but, le séquençage du génome d’A. baylyi a été réalisé au Genoscope puis annoté avec la participation d’experts du métabolisme bactérien (V. Barbe et al., 2004). Parmi les 3205 CDS (3) annotées, 36% ont une fonction connue, 28% une fonction " probable " et 36% n’ont pas de fonction associée. Cette première analyse a montré en autre que le génome d’A. baylyi plus simple que celui d’
E. coli renferme moins de duplications de gènes. Ceci devrait notablement simplifier l’analyse phénotypique de mutants. Un effort particulier a été apporté sur la reconstruction des voies métaboliques de cette bactérie.
Etablissement d’une collection de mutants de remplacement
L’analyse systématique s’est poursuivie par l’établissement d’une
collection de mutants de remplacement pour chaque CDS identifiée lors de l’annotation du génome. Sur les 3 205 CDS, 2594 mutants de remplacement ont été obtenus (81%). Nous n’avons pas pu obtenir de mutants avec une structure correcte pour 499 gènes qui constituent donc l’ensemble des candidats de gènes essentiels à la vie d’ADP1 en milieu minimum.
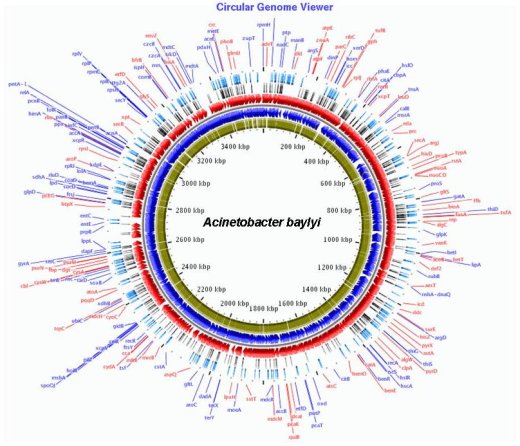
Représentation circulaire du génome d’A. baylyi. (Rouge et bleu) : gènes localisés sur le brin (+) et le brin (-). (Vert) : gènes pour lesquels un mutant a été obtenu. (Noir) : gènes pour lesquels aucun mutant n’a été obtenu. (Bleu clair) : enzymes appartenant à une voie de biosynthèse.
Glossaire :
(
1) Compétence (d’une bactérie) : capacité d’une bactérie à incorporer de l’ADN étranger.
(
2) Recombinaison homologue : processus d’échange entre deux régions d’ADN identiques.
(
3) CDS : Coding DNA Sequence.
Collection de mutants
Principe et Méthode :
Chaque CDS (
1), définie lors de l’annotation, a été remplacée par recombinaison homologue par une cassette contenant le gène de résistance à la kanamycine placé sous le contrôle d’un promoteur fort (promoteur du phage T5). Les extrémités de la cassette d’intégration, appelées "pieds de recombinaison", sont constituées de régions spécifiques qui encadrent le gène à muter. Le remplacement se fait en général sur la totalité de l’ORF(
2). Cependant, pour éviter l’altération de régions essentielles telles que le RBS (
3) ou le promoteur des gènes voisins du gène cible, nous avons été amenés à faire un remplacement en tronquant l’ORF cible de quelques acides aminés. La construction moléculaire des cassettes est obtenue
in fine par une PCR (
4) recombinante des pieds de recombinaison avec le gène de résistance à l’antibiotique. Après transformation d’
Acinetobacter baylyi par chacune de ces cassettes, les mutants sont sélectionnés sur un milieu minéral supplémenté en succinate et en kanamycine.
Technique :
La phase production :
L’optimisation du protocole de transformation ainsi que celle de l’organisation du " pipeline " nous a permis d’atteindre plus de 90% de réussite et une capacité de 400 mutants initiés toutes les deux semaines. Le cycle entier d’obtention d’un mutant depuis l’étape de transformation jusqu’à son stockage est de 4 semaines. Ce cycle inclut les PCR contrôles pour s’assurer du remplacement de l’ORF par la cassette d’intégration ainsi que le séquençage systématique des régions génomiques du mutant adjacentes à la cassette d’intégration.
Résultats :
Sur les 3 205 gènes annotés chez
A. baylyi et candidats à la mutation, nous avons tenté d’inactiver 3 167 gènes. Nous avons obtenu des mutants de remplacement pour 2 594 gènes, ce qui représente 81% des gènes d’
A. baylyi (
de Berardinis
et al., 2008).
Répartition des mutants obtenus selon la catégorie de fonction des gènes
Les données obtenues sur les gènes essentiels d’ADP1 sont cohérents à 88% avec celles obtenues sur Escherichia coli par la collection de mutants Keio profilée sur milieu minimum (Baba et al., 2006) ainsi qu’à 80% avec les données sur les orthologues de
Pseudomonas aeruginosa (Liberati et al., 2006).
Les CDS d’ADP1 ont été classés dans les catégories fonctionnelles de la classification TIGR. La distribution des gènes non essentiels et essentiels est présentée dans la figure ci-dessous.
Plusieurs stratégies ont été initiées pour explorer le métabolisme d’ADP1 :
- recherche des incohérences entre nos données d’essentialité et la connaissance actuelle du métabolisme,
- comparaison de nos données avec celles obtenues sur d’autres organismes,
- phénotypage systématique de la collection de mutants sur un ensemble de sources de carbone (quinate, glucose, 2-3 butanediol, ...).
- Cette collection représente donc une nouvelle ressource pour l’étude de la fonction des gènes et constitue un jeu de données expérimentales solides pour des approches de Systems biology (Durot
et al., en préparation).
Pour nous aider dans ces études, un
ADP1 Genome Browser a été mis en place dans le but de présenter les données de la collection de mutants dans leur contexte génomique : voies métaboliques KEGG et BioCyc, sous-système de SEED, prédictions d’opérons, gènes orthologues et données d’essentialité de plusieurs organismes, données de phénotypage systématique sur plusieurs sources de carbone...
Distribution des mutants de la collection
Un certain nombre de mutants ont déjà été distribuées et ont été utilisés dans plusieurs études (Chakravorty
et al., 2008 ; Aghaie
et al., submitted). La distribution des mutants est réalisée suivant le protocole
ci-dessous qui inclut les données sur les primers utilisés pour réaliser les délétions.
Perspectives :
La phase systématique de remplacement des gènes, maintenant achevée, sera prolongée par une phase de finition ciblée, pour essayer d’obtenir les mutants pour lesquels la première tentative a échoué. Une simple répétition des expériences devrait suffire pour les "échecs techniques" avec, pour un certain nombre d’entre eux, le choix de nouvelles amorces dans des régions plus favorables. Nous allons également essayer de répéter les expériences sur milieu plus riche afin d’obtenir les mutants des gènes qui sont essentiels sur succinate.
Une phase analytique de la collection va être maintenant abordée en plus du phénotypage systématique afin d’étudier, par exemple, des gènes de fonction inconnue co-localisés avec des gènes participant à la même voie métabolique.
Glossaire :
(
1)
CDS : Coding DNA Sequence.
(
2)
ORF : Open Reading Frame.
(
3)
RBS : Ribosome Binding Sequence.
(
4)
PCR : Polymerase Chain Reaction.
Demande de souches de mutants d’Acinetobacter baylyiADP1
- Toutes les souches sont gratuites (la collection complète n’est pas encore distribuée). Seuls les frais de transport sont à la charge du demandeur.
- Le transporteur utilisé pour les envois internationaux est FedEx
- Envoyez le document « request-strain-order » complété par mail à l’adresse suivante : adp1-mutants@genoscope.cns.fr
Un document MTA (Material Transfer Agreement) vous sera envoyé dans notre mail de confirmation de commande de mutants.
Le MTA rempli et signé par un responsable scientifique doit nous être retourné par courrier (et non par FAX ou mail) à l’adresse suivante :
Dr. Veronique de Berardinis
Genoscope/CEA
2, rue Gaston Crémieux
CP5706
91057 Evry Cedex
France
- Les souches seront préparées et envoyées après la réception du MTA
Les données sur les
primers utilisés pour la délétion des gènes ciblés ainsi que ceux utilisés pour faire les vérifications de délétion par PCR externes sont disponibles sous la forme d’un fichier au format .xls.