L'évolution des technologies de séquençage au cours des dernières années a révolutionné la génomique. La société Oxford Nanopore Technologies (ONT) commercialise des séquenceurs utilisant des nanoporesa, capables de lire des fragments d'ADN très longs, en général plusieurs dizaines de kilobases voir de l'ordre du mégabase pour les plus longues lectures. En s'appuyant sur ces technologies, plusieurs consortiums internationaux se sont attelés à établir des génomes de référence de très haute qualité : le consortium T2T (Telomere To Telomere) pour le génome humain ou encore le Vertebrate Genome Project (VGP) pour obtenir la séquence du génome de l'ensemble des vertébrés.
Le but de ces initiatives est d'obtenir la séquence complète des génomes étudiés. Bien que connue depuis environ 30 ans, la séquence du génome humain contient encore de nombreuses régions inconnues ou erronées (environ 8%). En 2020 le consortium T2T a proposé le premier génome humain complet.
Le séquençage et la reconstruction des génomes des plantes a toujours été considéré comme un challenge de par leur taille (un exemple : la taille du génome du pin qui équivaut à dix fois la taille du génome humain) et la présence de nombreuses séquences répétées. En effet, elles possèdent des séquences parfois de très grande taille et qui sont présentes un très grand nombre de fois dans leur génome. Ces séquences compliquent grandement la reconstruction complète de la séquence de leur génome. Cependant, l'évolution constante des technologies de séquençage et des outils informatiques permettent aujourd'hui d'envisager de reconstruire des génomes de plus en plus complexes avec une qualité jusque-là inatteignable.
C'est dans ce contexte que les chercheurs du Genoscope, en collaboration avec une équipe du CIRAD, ont réussi à reconstituer les séquences de chromosomes complets d'un génome de bananier (Musa acuminata, variété Pahang). Ces travaux ont fait l'objet d'une publication dans le journal Communications biology. Les bananiers sont des plantes contenant quatre grands groupes (Musa acuminata, Musa balbisiana, Musa schizocarpa, groupe des Australimusa) qui sont à l'origine de l'ensemble des espèces cultivées comestibles. Les cultures de bananiers sont particulièrement menacées par certaines maladies comme la cercosporiose noire (maladie responsable des tâches foliaires) qui nécessite un usage massif de pesticides, ou encore des infections par une nouvelle souche de Fusarium (champignon filamenteux) et pour lesquelles aucun contrôle chimique n'est possible. Il est donc indispensable pour les chercheurs d'avoir accès aux ressources génomiques les plus complètes pour étudier l'origine de ces espèces cultivées mais également pour rechercher des gènes de résistances aux pathogènes.
Le séquençage longue lecture réalisé sur le séquenceur PromethION d'Oxford Nanopore Technologies a permis de générer un assemblage du génome de très haute qualité. Pour la première fois, les chercheurs ont réussi à reconstruire la séquence complète, d'un télomère à l'autre, de plusieurs chromosomes et ceci en ne s'appuyant que sur une seule technologie. Des analyses complémentaires ont permis de révéler l'architecture très complexe de régions telles que les centromères ou les clusters de gènes codant pour les complexes ribosomaux. D'autre part, un focus sur les régions du génome contenant des gènes de résistance avec motifs NLR (motifs impliqués dans la reconnaissance de corps pathogènes) a mis en évidence une structure bien plus complexe que celle connue jusqu'à présent.
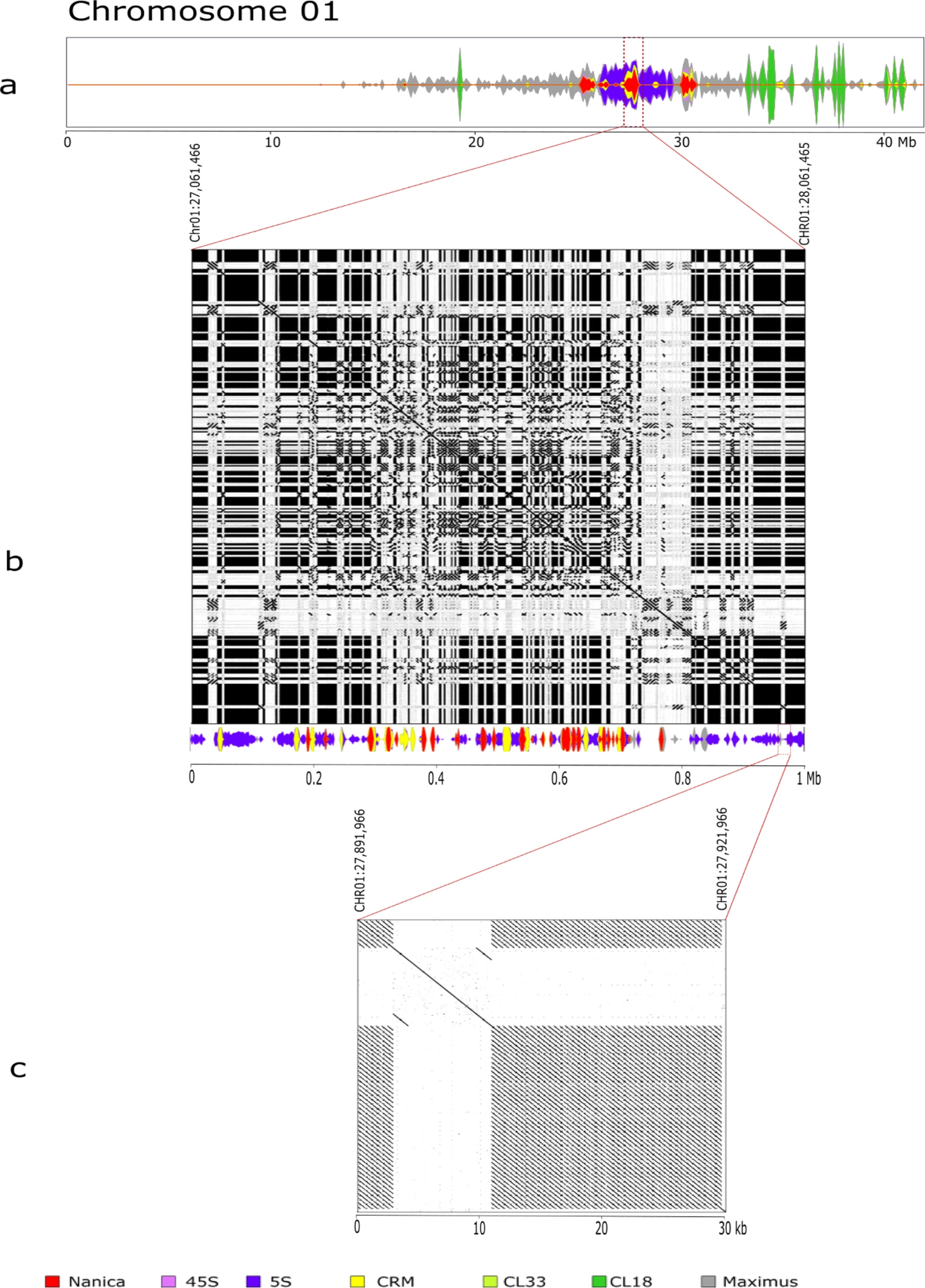
Principales séquences répétées péricentromériques du chromosome 1
a : Le séquençage par nanopores permet de déterminer l'ordre dans lequel les nucléotides sont disposés sur un fragment d'ADN donné à l'aide de pores à travers lequel passe un courant électrique. La quantité de courant qui peut passer dans le nanopore diffère dépendamment si le nanopore est obstrué par un nucléotide A, un T, un G ou un C ou encore d'une section d'ADN incluant plusieurs de ces bases. La quantité de courant qui traverse le nanopore permet de faire une lecture directe de la séquence d'ADN.