L'accélération des progrès dans les technologies de séquençage donnent le vertige. En 2003, les scientifiques sont venus à bout d'un génome humain après 15 ans de travail et pour 2 milliards d'euros. Aujourd'hui, les industriels proposent des appareils permettant de séquencer un génome pour moins de 1000 euros en quelques heures seulement. Toutefois, ces technologies « courtes lectures » ne permettent pas de résoudre les problèmes d'assemblage des génomes complexes (comprenant de nombreuses séquences répétées). Les technologies de séquençage « longues lectures » ont été lancées il y a quelques années. Elles permettent, contrairement aux technologies courtes lectures, de résoudre les problèmes d'assemblage des génomes grands et complexes. Cependant, ces technologies possèdent encore des limitations, notamment un taux d'erreur élevé (15-30%), compliquant leur utilisation par les laboratoires de recherche. Des chercheurs du CEA-IG ont eu l'idée de combiner les deux approches avec une technique accessible sur le marché et l'autre encore en période de tests. Cette méthode deux-en-un permet de séquencer et d'obtenir la séquence complète du génome de bactéries avec une bonne fiabilité et en utilisant des séquenceurs faciles à mettre en œuvre et peu onéreux. La méthode et les données sont mises à la disposition de la communauté scientifique sur le site du Génoscope (http://www.genoscope.cns.fr/nas).
 « L'astuce consiste à combiner les données provenant d'un appareil précis générant de courtes lectures avec des longues lectures imparfaites », explique Jean-Marc Aury, chercheur au CEA-IG. Ces longues lectures sont obtenues avec l'appareil MinION® de la société Oxford Nanopore Technology. De la taille d'une clé USB, cet outil est testé actuellement par un ensemble de laboratoires dont le Génoscope. La lecture courte provient d'un appareil classique de la taille d'un ordinateur, commercialisé par la société Illumina. Ainsi, telle la reconstitution d'un puzzle complexe avec des pièces similaires difficiles à placer, la lecture longue donne une vision globale, mais « floue », du génome. La lecture courte donne quant à elle le motif précis des pièces du puzzle. « Nous alignons les courtes lectures sur les plus grandes, poursuit le scientifique. La lecture longue permet de positionner les motifs du génome qui se répètent. Les tests sur la bactérie modèle Acinetobacter baylyi ADP1 ont affiché 95 % de lecture sans erreur. Nous avons ainsi obtenu une lecture de 91 kilobases sans erreur. » Lancés en parallèle, les deux séquenceurs donnent leurs résultats en moins de deux jours. Moins d'une journée suffit ensuite pour reconstituer l'assemblage du génome. Si le génome de cette bactérie détient 1000 fois moins de bases que celui de l'Homme (3.6 Mégabases versus 3 Gigabases), cette méthode constituera un moyen pour les laboratoires de séquencer leurs modèles de recherche pour un coût limité et sur un coin de leur paillasse.
« L'astuce consiste à combiner les données provenant d'un appareil précis générant de courtes lectures avec des longues lectures imparfaites », explique Jean-Marc Aury, chercheur au CEA-IG. Ces longues lectures sont obtenues avec l'appareil MinION® de la société Oxford Nanopore Technology. De la taille d'une clé USB, cet outil est testé actuellement par un ensemble de laboratoires dont le Génoscope. La lecture courte provient d'un appareil classique de la taille d'un ordinateur, commercialisé par la société Illumina. Ainsi, telle la reconstitution d'un puzzle complexe avec des pièces similaires difficiles à placer, la lecture longue donne une vision globale, mais « floue », du génome. La lecture courte donne quant à elle le motif précis des pièces du puzzle. « Nous alignons les courtes lectures sur les plus grandes, poursuit le scientifique. La lecture longue permet de positionner les motifs du génome qui se répètent. Les tests sur la bactérie modèle Acinetobacter baylyi ADP1 ont affiché 95 % de lecture sans erreur. Nous avons ainsi obtenu une lecture de 91 kilobases sans erreur. » Lancés en parallèle, les deux séquenceurs donnent leurs résultats en moins de deux jours. Moins d'une journée suffit ensuite pour reconstituer l'assemblage du génome. Si le génome de cette bactérie détient 1000 fois moins de bases que celui de l'Homme (3.6 Mégabases versus 3 Gigabases), cette méthode constituera un moyen pour les laboratoires de séquencer leurs modèles de recherche pour un coût limité et sur un coin de leur paillasse.
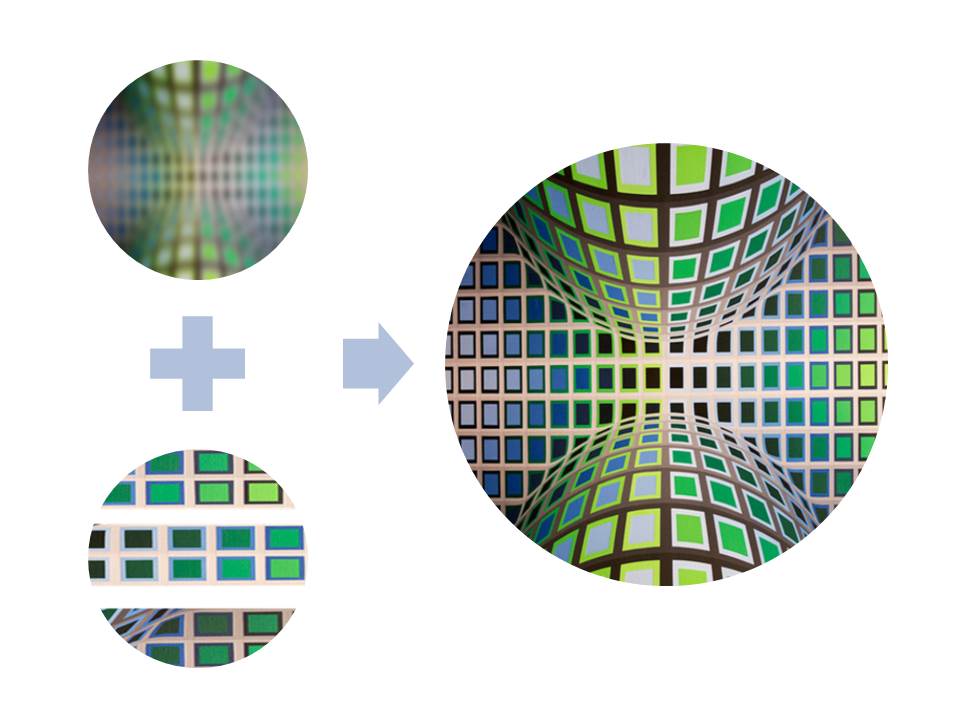
Représentation imagée de la combinaison des deux techniques de séquençage. A gauche : la longue lecture donne une image globale mais approximative (en haut), la courte lecture propose des échantillons de bonne définition (en bas). A droite : la somme des courte et longue lectures permettent de visualiser l'ensemble avec une bonne définition. (c)E. Varrier & CEA